El VIH fue descubierto y considerado como el agente de la naciente epidemia de
sida por el equipo de
Luc Montagnier en
Francia en 1983. El
virión es esférico, dotado de una envoltura y con una cápside proteica. Su genoma es una cadena de
ARN monocatenario que debe copiarse provisionalmente al
ADN para poder multiplicarse e integrarse en el genoma de la
célula que infecta. Los antígenos
proteicos de la envoltura exterior se acoplan de forma específica con proteínas de la membrana de las células infectables, especialmente de los
linfocitos T CD4.
Desde su ingreso en la célula hospedadora, la cadena simple de ácido ribonucleico (
ARN) viral comienza su transformación en una doble cadena de ácido desoxirribonucleico (
ADN) por acción de la
enzima transcriptasa inversa que forma parte del virus. La
integrasa y otros cofactores actúan para que el ARN del virus se fusione con el ADN de la célula hospedadora
12 a través de la
transcripción en el genoma de la célula que aloja al virus. De esta manera, la célula queda infectada por el virus. Después de este proceso, los lentivirus reaccionan de dos maneras posibles: puede ocurrir que el virus entre en
latencia mientras la célula infectada continúa en funciones, o bien, que el virus comience a replicarse activamente y libere viriones capaces de infectar otras células.
Existen dos tipos del VIH, llamados VIH-1 y VIH-2. El primero de ellos corresponde al virus descubierto originalmente, que recibió los nombres de LAV y HTLV-III por parte de los dos equipos que estaban investigando el agente etiológico del sida durante la primera mitad de la
década de 1980. El VIH-1 es más virulento e infeccioso que el VIH-2
13 y es el causante de la mayoría de infecciones por VIH en el mundo. El VIH-2 es menos infeccioso y se encuentra confinado casi exclusivamente a los países de
África occidental.
14
Estructura y genoma del VIH
Estructura
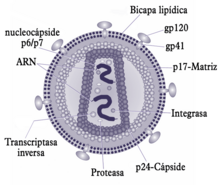
Estructura del VIH.
El VIH comparte con los
retrovirus las características esenciales de esa familia. El
virión contiene información genética bajo la forma de ácido ribonucleico (
ARN), protegido por una envoltura de membrana. Los retrovirus insertan su información genética en las células hospedadoras por acción de la
transcriptasa inversa.
15
Un virión del VIH tiene una forma aproximadamente esférica con un diámetro de 80-100
nm. Está constituido por tres capas. La exterior es una bicapa
lipídica. Posee 72 espículas formadas por las
glicoproteínas gp120 y
gp41 que actúan en el momento de la unión del virus a la célula hospedadora. La capa intermedia está constituida por la núcleo
cápside icosaédrica. La capa interior tiene forma de un cono truncado. Está constituida por el ARN viral y la nucleoproteína. La cadena genética del VIH está constituida por un
ARN de cadena simplecompuesto por dos filamentos idénticos. El ARN contiene varios genes, cada uno de los cuales codifica las diversas proteínas que el VIH necesita para reproducirse.
16
Genoma y composición
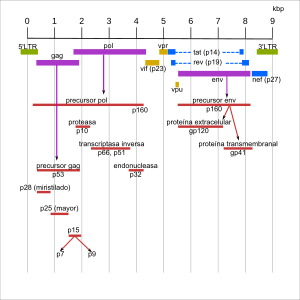
Genoma del VIH-1.
Los genomas del VIH-1 y VIH-2 son muy similares. Ambos están compuestos por los tres genes básicos de la familia de los retrovirus. Se trata de los genes
gag,
pol y env. Cada uno de estos genes codifica
proteínas que ayudan a la reproducción del virus. El genoma del VIH posee otros seis genes adicionales:
tat,
vpr,
rev,
vpu (
vpx en el caso del VIH-2),
vif y
nef.
17
Genes estructurales
Las proteínas estructurales son codificadas por los genes
gag,
pol y
env, y su secuencia cubre la mayor parte del genoma viral, quedando solo una parte menor para el resto de los genes.
El
gen gag es traducido a una proteína precursora, la p55, que luego se asocia, durante la gemación por la que se liberan nuevas partículas víricas desde la célula infectada, a dos copias del ARN viral, para el que presenta una región afín, y a otras proteínas virales y celulares. Una
proteasa, producto del gen
pol corta durante la maduración del virión la p55 en cuatro proteínas que se incorporan a sus lugares respectivos:
- La proteína p24 forma la cápside.
- La proteína p17 constituye la matriz, situada bajo la envoltura, a la que estabiliza. Una parte de las proteínas se unen al complejo molecular que acompaña al ADN viral al interior del núcleo. En la superficie de la proteína existe una región cariofílica (literalmente afín al núcleo) que es reconocida por la maquinaria molecular de importación nuclear. Este es el mecanismo que permite al VIH infectar células diferenciadas, no destinadas a dividirse, algo que no ocurre en ningún otro retrovirus.
- Las proteínas p6 y p7 (o p9) forman la nucleocápside. La región de la p55 correspondiente al polipéptido p6 es responsable de la incorporación de la proteína accesoria Vpr (producto de la traducción del gen vpr) al virión en formación y de la interacción con la membrana de la célula que hace posible la gemación. La p7 (p9) es responsable del reconocimiento y la incorporación del ARN al virión y además interviene en la transcripción inversa facilitándola.
Dentro de la cápside, además de las dos copias idénticas del ARN viral hay ejemplares de tres enzimas necesarias para la multiplicación del virus: una transcriptasa inversa, una integrasa y una proteasa. Estas enzimas, así como una ARNasa se producen a partir de la proteína Pol, después del corte de una proteína precursora mixta derivada de la cotraducción, una de cada 20 veces, de los genes
gag y
pol. La propia proteasa vírica rompe la proteína anterior, con una eficiencia limitada, para obtener las proteínas Gag (p55) y Pol. Luego la proteína precursora Pol es cortada a su vez para formar las cuatro proteínas funcionales citadas:
- La proteasa (p10). Se trata de una aspartil-proteasa cuya forma funcional es un dímero del que se conoce la estructura tridimensional. Actúa cortando las piezas de las proteínas Gag, Pol y de la Gag-Pol. Una parte de los fármacos empleados contra el VIH son inhibidores de su función.
- La transcriptasa inversa (p50) cuya función es la síntesis del ADN de doble cadena del provirus usando como patrón la cadena singular del ARN viral. Es una ADN-polimerasa que puede actuar como dependiente del ADN tanto como del ARN. Una vez formada la primera cadena de ADN, complementaria del ARN viral, la ARNasa lo separa de él, lo que permite a la transcriptasa inversa ejecutar la síntesis de la segunda cadena de ADN tomando como molde la primera que se formó. Así pues, para la síntesis de la primera cadena la actividad de la transcriptasa inversa es ARN-dependiente, pero para la de la segunda es ADN-dependiente. También existen múltiples fármacos contra la actividad de la transcriptasa inversa.
- La ARNasa (p15), que como se ha dicho separa las cadenas de ARN de las de la ADN durante la transcripción inversa.
- La integrasa(p31) realiza la inserción del ADN proviral en el genoma de la célula huésped. No se requiere ATP para su actividad y debe cumplir sucesivamente tres funciones:
- Con una actividad exonucleasa corta dos núcleótidos del extremo 3' de cada una de las dos cadenas del ADN proviral.
- Con una actividad endonucleasa (de doble cadena) corta el ADN del huésped en el punto de integración. No hay un lugar fijo en el genoma para que esto se realice, sino que ocurre en cualquier región muy accesible de la cromatina, lo que se supone que favorece la expresión del provirus, al coincidir esas regiones del genoma con las más transcritas.
- Por último, con una actividad ligasa el ADN proviral es soldado, mediante solo un enlace covalente en cada extremo, en el ADN celular.
En la actualidad existe un fármaco comercializado
contra la actividad de la integrasa, el
raltegravir.
La envoltura se basa en una bicapa lipídica, lo mismo que cualquier membrana biológica, y sus componentes estructurales básicos proceden de la membrana plasmática de la célula parasitada. Pero la envoltura porta además regularmente espaciadas 72 espículas, que son complejos proteicos integrados en la membrana formados por proteínas virales codificadas por el gen
env. Cada espícula está formada por una pieza de la proteína gp41, integral en la membrana, y una cabeza externa formada por la proteína gp120, esencial para el acoplamiento con el exterior de ciertas células previo a su invasión. Entre los dos componentes de las espículas existe una unión no
covalente. Las proteínas gp41 y gp120 se sintetizan como una sola poliproteína, gp160, con la información del gen
env antes de que sea cortada por una proteasa de la célula. La proteína Env existe como trímero en la superficie de los viriones y las células infectadas.
Los
fármacos inhibidores de la fusión funcionan contra la proteína gp41, para evitar su unión a los
linfocitos.
Proteínas reguladoras
Tat
La proteína
Tat existe en dos formas, una larga, de 101 restos aminoácidos de longitud, y otra más corta, de sólo 72. La segunda se produce cuando en fase temprana se produce una edición completa del ARNm viral, la primera cuando en una fase más tardía solo se realiza una edición parcial. La proteína Tat (por
transactivator) es imprescindible para la producción de nuevos viriones, que promueve activamente. La proteína se une a una región de 59 nucleótidos situada en el extremo 5' del ARN viral llamada TAR (
Transactivator Active Region) y actúa como un transactivador, algo excepcional, puesto que éstos suelen unirse al ADN, no al ARN. En cuanto este extremo inicial del genoma viral ha sido transcrito desde el ADN proviral, la proteína Tat se une a él y promueve su elongación favoreciendo la transcripción del resto de la cadena.
Rev
La proteína
rev regula la expresión del ARN viral controlando el ritmo de exportación del ARNm.
18
Tat y Rev: acción conjunta
La acción sinergística de Tat y Rev fuertemente incrementa la expresión de proteínas virales. Los papeles que Tat y Rev desempeñan en la regulación transcripcional del VIH-1 y en la expresión de proteínas estructurales, respectivamente, hacen Tat y Rev esenciales para el ciclo de vida del VIH. Sus funciones facilitan la expresión de proteínas virales en dos etapas. Después de la integración del ADN proviral y de su transcripción en un nivel basal, solamente los RNAms de 2 KB se transportan al citoplasma. Esto permite la síntesis de Tat, Rev y de Nef. Tat y Rev entonces son transportadas al núcleo, donde actúan para aumentar la transcripción del ADN del provirus (Tat) y del transporte de todos los RNAms virales al citoplasma (Rev). La expresión de proteínas codificada por las clases de RNAm de 9 KB y 4 KB (Gag, Gag-Pol, Env, Vpr, Vif, y de Vpu) puede entonces ocurrir. Estudios donde se han mutado genes virales han determinado que Vif, Vpr, Vpu y Nef no son esenciales para la producción de partículas infecciosas en cultivos celulares "in-vitro". Sin embargo, la conservación de dichas proteínas accesorias en el genoma del VIH sugiere que todas desempeñan papeles importantes durante el ciclo infeccioso en el huésped. Los roles de estas proteínas serán descritos a continuación.
Proteínas accesorias
Vif: incremento en infectividad y protección del genoma viral
Vif es una proteína de 193 aminoácidos que está presente en bajos niveles adentro de los viriones, e interactúa con en RNA genómico viral. La división de esta proteína reduce la infectividad del VIH-1 en cultivos celulares y en modelos animales de patogénesis. No obstante, el mecanismo de acción de Vif se ha empezado a entender recientemente. La ausencia de Vif en partículas infecciosas no puede ser compensada con la expresión de Vif en las células infectadas. Estudios recientes han demostrado que Vif es requerida para eliminar la acción del factor ApoBEC3G, la cual es una deaminasa de citidinas, que convierte la citosina en uracilo, y emplea como sustrato el
ADN de cadena sencilla. Además, esta enzima posiblemente actúa durante el ciclo de la transcriptasa inversa, modificando así la cadena negativa del DNA, porque esta es la fase en la cual el ADN de cadena sencilla está disponible. ApoBEC3G es selectivamente incorporada dentro de las partículas de VIH, resultando en un alto nivel de mutaciones en el genoma viral. Dado que estos altos niveles de mutación son perjudiciales para la viabilidad del virus, VIH ha evolucionado una estrategia para abolir esta poderosa barrera. Sin embargo, estudios recientes sugieren que ApoBEC3G no requiere su acción enzimática para tener efecto. Estudios más recientes han implicado que ApoBEC3G tiene un rol en la inhibición de ciertas fases en el ciclo de la transcriptasa inversa.
Vpu: facilita el desprendimiento de viriones en células infectadas
Vpu es una proteína de 81 aminoácidos que es insertada en membranas vía su terminal nitrogenado. Vpu se acumula en el aparato de Golgi y en endosomas celulares. Vpu es única en HIV-1 y no hay homólogos en lentivirus relacionados como el VIH-2 y el VIS. A Vpu se le han atribuido dos actividades.
Degradación de la proteína CD4
En la ausencia de Vpu, la proteína CD4 interactúa con la proteína viral gp160 recién sintetizada para formar un complejo insoluble, el cual retiene gp120 dentro de la
célula. La región citoplásmica de Vpu se puede unir con CD4 y con la proteína β-TrCP. Esto induce la ubiquitinización de CD4 y su subsiguiente degradación por el
proteasoma, incrementando así la expresión de gp120 en la superficie celular.
Realza en el desprendimiento de viriones de la membrana celular
Esta actividad depende de la región transmembranal de Vpu. En la ausencia de Vpu, los viriones se acumulan en la superficie celular en un estado parcialmente desprendido. Expresión de Vpu resulta en la liberación facilitada de viriones de la membrana celular. Remarcablemente, este efecto no está restringido solamente al VIH-1; Vpu también facilita el desprendimiento de otros virus no relacionados. El mecanismo por la cual esto ocurre es desconocido. Se ha sugerido que Vpu facilita la fluidez de la membrana celular por medio de un canal de cationes. También se ha sugerido que Vpu causa disrupción de interacciones entre proteínas del VIH y de la superficie celular; esto previene la endocitosis de viriones recientemente desprendidos de la célula...


 agenciaajn.com
agenciaajn.com